Remarques:
1) Ce protocole remplace les versions précédentes (PCR locus D1S80 et PCR locus PV92). Ces expériences peuvent toutefois être encore empruntées sur demande.
2) Ce nouveau protocole a été mis en place car il permet de faire le lien entre un génotype donné et le phénotype associé. L'expérience se déroule en deux étapes/séances: la première séance est consacrée à l'extraction d'ADN, à la PCR et au test de détection du goût amer (test PTC) et la deuxième séance à la digestion, à la migration sur gel d'agarose et à l'analyse des résultats. Les échantillons peuvent être conservés à -20°C entre les deux séances.
Un atelier de bioinformatique intitulé "La génétique du goût amer" en lien avec cette expérience a été mis en place par Marie-Claude Blatter du SIB Institut Suisse de Bioinformatique et peut être visualisé ici. Cet atelier constitue un joli complément à l'expérience de PCR.
Information importante au sujet de la sécurité des bandelettes PTC:
L'utilisation de ces bandelettes relève de la responsabilité de l'enseignant, merci de prendre connaissance de la feuille d'information du fournisseur ci-dessous.
Information au sujet de la toxicité des bandelettes PTC (traduction en français)
Information regarding Carolina Biological PTC Papers (document original en anglais)
La PCR:
La technique de polymérisation en chaîne (en anglais « polymerase chain reaction ») ou PCR, permet d’amplifier des millions de fois un unique fragment d’ADN. Cette méthode est devenue un outil précieux non seulement pour la recherche en biologie moléculaire mais également pour le diagnostic médical, la détermination de microorganismes ou encore la criminologie. L’invention de la PCR revient à Kary Mullis dans les années 80. Il obtint pour cette découverte le Prix Nobel de Chimie en 1993. La découverte d’ADN polymérases thermorésistantes (la Taq polymérase par exemple) isolées de bactéries (Thermus aquaticus) vivant dans des sources d’eau chaude a facilité l’utilisation de la PCR et a permis son automatisation.
Thèmes : la PCR, la structure du génome humain, le génotypage, la criminologie, l’ADN, l’électrophorèse.
Une réaction de PCR nécessite :
- de l’ADN à amplifier (dénommé template)
- des amorces (également appelés primers)
- des desoxynucléotides triphosphates (dATP, dCTP, dGTP, dTTP)
- une ADN polymérase thermorésistante (la Taq par exemple)
- du magnésium (Mg++) indispensable au fonctionnement de l’ADN polymérase
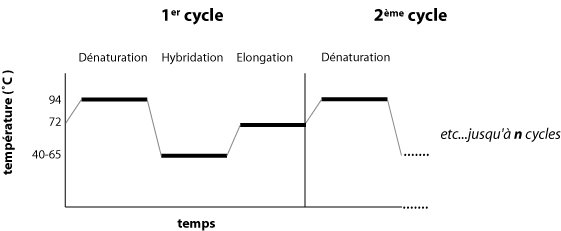
Ces 5 ingrédients sont mélangés dans un tube à essai et soumis à différents cycles de températures.
 |
 |
1ère étape : la dénaturation (94°C). 2ème étape : l’hybridation (40-65°C). 3ème étape : l’élongation (72°C). |
Ces trois étapes correspondent à 1 cycle de PCR, à la suite duquel on a doublé le nombre de fragment d’ADN cible initial. Environ une trentaine de cycles sont en principe effectués dans une expérience de PCR classique. Ainsi, après 30 cycles on aura amplifié 2n fois notre ADN cible.
Un schéma de l'amplification par PCR est visible ici:
Protocole PCR sensibilité au PTC
L’homme est capable de distinguer 5 saveurs fondamentales, le sucré, le salé, l’acide, l’amer et l’umami (glutamate) grâce à des récepteurs situés au niveau de bourgeons gustatifs.
Dans les années 30, un chimiste du nom d’Arthur Fox est le premier à mettre en évidence la différence de sensibilité au PTC (phénylthiocarbamide), un composé organique amer produit par diverses plantes comme le brocoli. Alors qu’il venait de synthétiser ce composé, l’un de ses collègues s’est plaint de l’amertume du produit alors que lui ne sentait rien.
Quelques années plus tard, une étude menée par Albert Blakeslee a montré que l’incapacité à sentir le PTC est un trait génétique récessif qui varie au sein de la population. Les individus sensibles peuvent détecter le PTC (allèle T+) et les individus non sensibles ne peuvent pas le détecter (allèle t-).
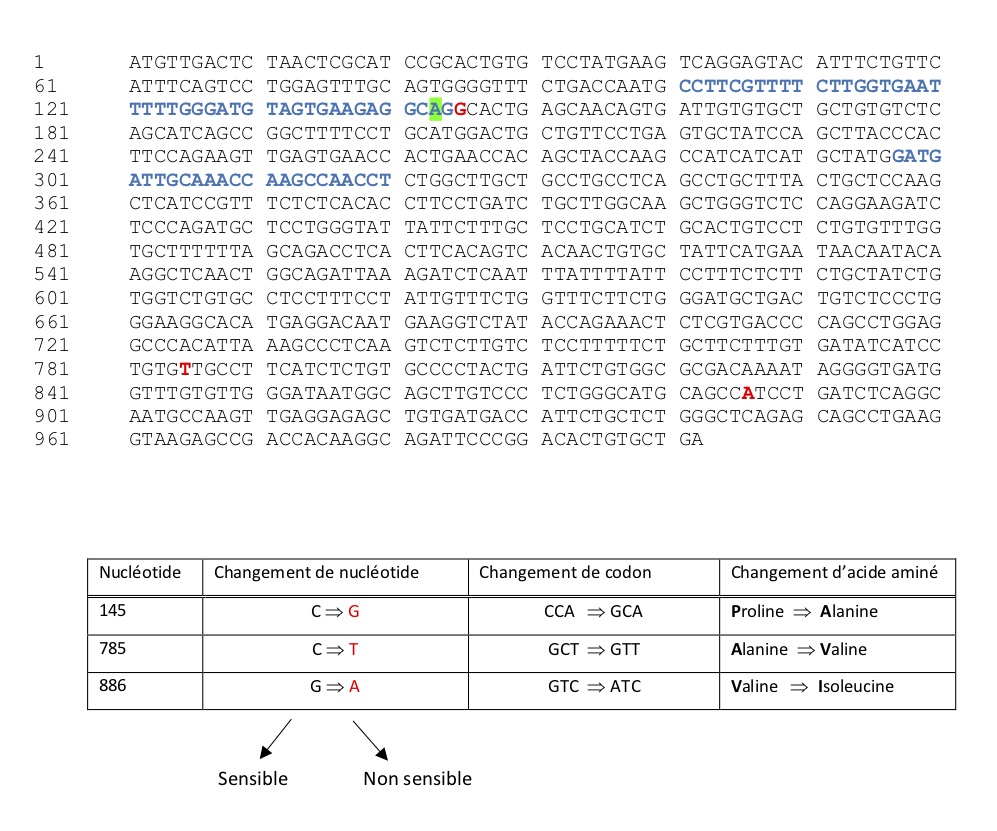
Il existe une trentaine de récepteurs sensibles aux différentes substances amères. Identifié en 2003, le gène TAS2R38, localisé sur le chromosome 7, code pour le récepteur au PTC. Le séquençage de ce gène a permis de mettre en évidence la présence d’une variation génique entre les individus sensibles et non sensibles. Cette variation se caractérise par une différence de 3 nucléotides, ou SNPs, dans le gène TAS2R38 (en rouge sur la séquence du gène ci-dessous).
SNP
Un SNP (Single Nucleotide Polymorphisme) est une variation dans une séquence d’ADN qui arrive lorsqu’un seul nucléotide diffère entre les membres d’une même espèce. Ces variations sont très fréquentes dans le génome humain, environ 1/1000. Elles représentent d’ailleurs 90% de l’ensemble des variations génétiques humaines.
Gène TAS2R38 ( individu non sensible au PTC, allèle t- ) :
Pour accéder à cette séquence en format texte, cliquez ici
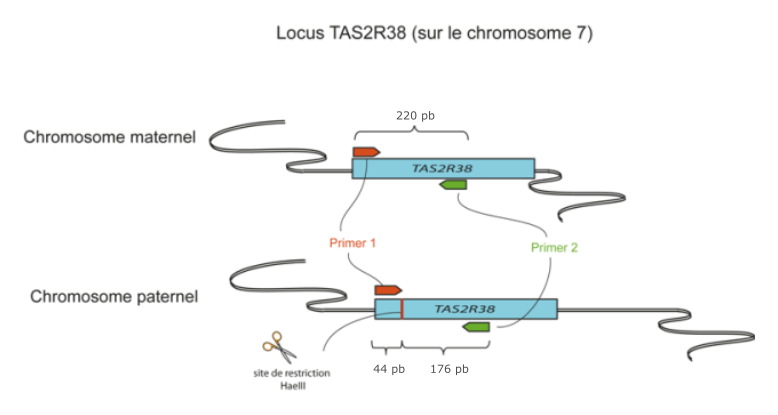
Primer sens 5’ CCT TCG TTT TCT TGG TGA ATT TTT GGG ATG TAG TGA AGA GGC GG 3’
Primer anti-sens 5’ AGG TTG GCT TGG TTT GCA ATC ATC 3’
Note: Le primer sens a été conçu de manière à obtenir le site de restriction de l'enzyme HaeIII, càd 5' GGCC 3' dans le cas d'un individu sensible au PTC.
L'EXPERIENCE:
Protocole :
1) Extraction d'ADN à partir de cellules épithéliales de la bouche
- Mettre 500 µl d'H2O dans un tube Eppendorf
- Frotter abondamment l’intérieur des joues avec un écouvillon (coton tige) stérile afin d’obtenir un maximum de cellules épithéliales
- Tremper l'écouvillon dans le tube contenant les 500 µl d'H2O. Tourner l'écouvillon et le frotter contre les parois du tube
- Bien essorer l'écouvillon en le sortant du tube
- Centrifuger le tube 2 minutes à la vitesse maximum afin de faire tomber les cellules épithéliales.
- A l’aide de la P1000 enlever le surnageant en prenant soin de laisser le culot de cellules au fond du tube. Attention à ne pas aspirer le culot de cellules. Le cas échéant centrifuger à nouveau la salive.
- Ajouter 0.1 ml de solution de NaOH 200 mM sur le culot et le resuspendre en vortexant ou en pipetant à l'aide de la P1000
- Fermer le tube et incuber à 95 °C pendant 10 minutes dans le bloc chauffant
- Vortexer brièvement
- Ajouter 0.1 ml de HCl 200 mM dans votre tube.
- Ajouter 0.1 ml Tris-HCl (pH 8.5) 200 mM dans votre tube.
- Votre préparation d’ADN est prête et peut être stockée au congélateur ou utilisée directement pour la suite de l’expérience
2) Amplification par PCR
- Préparer le mélange de réactif pour la PCR juste avant l'emploi. Ce mélange contient le 2 x Taq mix (taq polymérases, dNTPs, tampon de polymérisation et tampon de charge rouge), les primers et de l’H2O.
- Pour une classe de 16 élèves par exemple, on compte 16 réactions plus un contrôle négatif ainsi qu'un contrôle positif soit un total de 18 réactions. Afin de faciliter le pipetage et pour avoir assez de matériel en cas d'erreurs nous prévoyons un mélange pour 24 réactions.
| 1 réaction | 24 réactions | |
| 2xTaq ReadyMix | 12.50 µl | 300 µl |
| Primers mix PTC | 5.00 µl | 120 µl |
| DMSO | 1.25 µl | 30 µl |
| H2O | 1.25 µl | 30 µl |
- Ce mélange est préparé dans un tube Eppendorf 1.5 ml juste avant l’emploi. Mélanger en pipetant en haut et en bas délicatement.
- Distribuer ensuite les 20 µl du mélange dans les petits tubes de PCR préalablement marqué sur le côté par les initiales des personnes à tester.
- Dans votre petit tube ajouter 5 µl de votre solution d’ADN. Fermer le tube.
- Dans le petit tube PCR destiné au contôle négatif, ajouter 5 µl d’H2O à la place de l’ADN. Fermer le tube.
- Dans le petit tube PCR destiné au contôle positif, ajouter 5 µl d’ADN B2B. Fermer le tube.
- En suivant les instructions du mode d'emploi de la machine PCR, mettre les tubes dans la machine et lancer le programme PTC.
- Le programme dure environ 1h30. Il comporte les cycles suivants:
| 94° 5 minutes | ||
| 94° 30 secondes | } | |
| 64° 45 secondes | 30 cycles | |
| 72° 45 secondes | ||
| 72° 5 minutes |
- A la fin de la réaction de PCR les tubes peuvent être gardés au congélateur
- Arrêter la machine PCR en suivant les indications du mode d’emploi.
3) Digestion du produit PCR
- Transférer 20µl du produit PCR dans un tube eppendorf.
- Ajouter 5µl du mix de digestion
|
1 x
|
22 x
|
||
| Tampon digestion 10x |
2.5µl
|
55µl
|
|
| Enzyme HaeIII |
1 µl
|
22 µl
|
|
| H2O | 1.5 µl | 33µl |
- Fermer le tube, le centrifuger quelques secondes afin de tout faire tomber au fond du tube.
- Placer votre tube eppendorf à 37° et laisser digérer 20-30minutes.
- A la fin de la digestion, les tubes peuvent être gardés au congélateur pour la leçon suivante.

4) Préparation du gel d’agarose
Le TBE 10x concentré doit être manipulé uniquement par les enseignants ou les préparateurs, en prenant soit de porter une blouse, des lunettes et des gants en nitrile. Le TBE 1x concentré peut sans problème être manipulé par les élèves, à conditions qu’ils respectent les mêmes conditions de sécurité.
Le TBE est toxique pour l’environnement, il ne faut donc pas le jeter à l’évier. Celui-ci doit être récupéré et éliminé par le biais de votre propre filière de traitement des produits chimiques.
Le gel d'agarose est une matrice pour séparer les molécules d'ADN selon leur taille dans un champ électrique. Les petits fragments migrent plus vite que les grands, la concentration d'agarose est choisie en fonction de la taille des fragments à séparer. Ici nous allons utiliser un gel à 2% d’agarose.
- Peser 1.4 g d'agarose et mettez-le dans un Erlenmeyer de 250ml.
- Ajouter 70 ml de tampon d'électrophorèse (TBE 1x).
- Faire bouillir (four à micro-ondes, plaque chauffante ou bec bunsen). Veillez à ne pas que l’agarose déborde en bouillant ! L’agarose fondu est complètement transparent. Au cas où des résidus sont toujours visibles, remettre la solution à chauffer.
- Laisser refroidir le liquide (à environ 60°C).
- Ajouter 7 µl de SYBR-safe (intercalant qui permettra de visualiser l’ADN) et mélanger en agitant l’Erlenmeyer.
- Verser l'agarose dans la cuve préparée avec un peigne pour former les puits (attention si l'agarose est trop chaud la cuve d'électrophorèse se déforme !)
Les cuves d'électrophorèse possèdent des petits blocs en plexi pour protéger les électrodes. Souvent les cuves sont déformées et les blocs ne rentrent plus. Il suffit donc de couper, après solidification du gel, une petite lamelle d’agarose en haut et en bas du gel pour libérer les électrodes.
- Lorsque le gel est refroidi et solidifié ajouter du tampon d'électrophorèse (TBE 1x) et enlever le peigne.
5) Analyse des génotypes sur gel
- Récupérer vos tubes. Ils contiennent déjà le tampon de charge (rouge) et sont donc prêt à être mis sur gel.
- Charger le gel:
1er puits: 5 µl de marqueur.
2ème puits et suivants: 20 µl de vos échantillons.
Avant dernier puits: le contrôle négatif.
Dernier puits: le contrôle positif.
- Allumer le transformateur et faire migrer à 100V (ampérage maximum).
- Quand le colorant rouge de migration arrive aux trois quarts du gel, arrêter le transformateur.
- Laisser refroidir le gel quelques instants. Eventuellement en le mettant au frigo. L’intensité du signal sera ainsi plus forte.
- Prendre le gel et visualiser les bandes sous la lampe bleue.
- Prendre une photo.
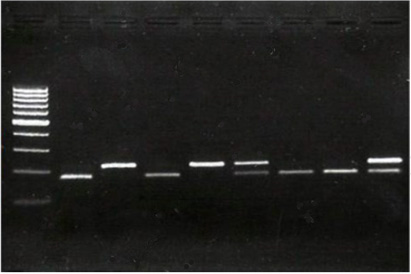
6) Résultats et discussions
Voici un exemple de résultat :
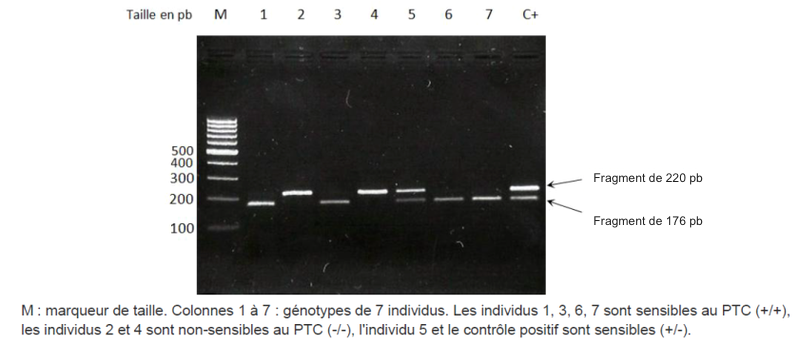
Homozygote non sensible (-/-), c'est à dire qui ne peut pas détecter le goût amer du PTC: 1 fragment de 220 bp
Homozygote sensible (+/+), c'est à dire qui peut détecter le goût amer du PTC: 2 fragments de 176 et 44 bp
Hétérozygote (sensible, +/-), qui peut également détecter le goût amer du PTC: 3 fragments de 220, 176 et 44 bp
Remarque: la bande à 44 bp n’est pas visible ici.
Analyse du génotype
Observez votre génotype et ceux des autres personnes.
- Calculez la fréquence des génotypes de la classe.
- Calculez la fréquence des allèles de la classe.
- Calculez la valeur de Hardy-Weinberg pour la classe.
Un autre exemple d'analyse des résultats (expérience sur le locus PV92) avec calcul de l'équilibre Hardy-Weinberg est accessible ici
Analyse du phénotype
La sensibilité au PTC est un trait génétique dominant. Ainsi les personnes homozygote (+/+) et hétérozygote (+/-) devraient détecter l’amertume du produit. Pour le vérifier, vous allez recevoir deux papiers, l’un imprégné de PTC et l’autre pas.
Commencez par déposer sur votre langue le papier contrôle, non imprégné et testez ensuite le papier imprégné de PTC.
Que constatez-vous ?
Vérifiez s'il y a concordance entre le génotype et le phénotype.
Matériel fourni:
- 2x Taq ReadyMix
- Primers mix
- DMSO
- ADN contrôle B2B
- Marqueur de taille (électrophorèse)
- Solution de SYBR-safe
- NaOH 200mM
- HCl 200mM
- Tris-HCl 200mM
- Tampon de digestion
- Enzyme HaeIII
- Agarose
- Boite tubes Eppendorf 1,5 ml
- Boite tubes PCR
- Portoir tube PCR
- Portoir tube eppendorf
- Micropipettes P20
- Micropipettes P200
- Micropipettes P1000
- Boîte de pointes jaunes
- Boîte de pointes bleues
- Cuve d'électrophorèse
- Lampe bleue
- Machine PCR
- Bloc chauffant
- Mini-centrifugeuse
- Ecouvillons
- Tampon d'électrophorèse (TBE 1x)
- Bouteille d'eau stérile
- Bandelette PTC / C -
Matériel non fourni:
L’Université de Genève décline toutes responsabilités en cas de dommages survenus durant les expériences.