Cette expérience est le fruit d'une collaboration avec le Bioscope et complète l'activité "Code my plant" proposée par ce dernier.
C’est une technique de taxinomie moléculaire permettant d’identifier un organisme grâce à un court marqueur génétique présent chez tous les êtres vivants. Les différences dans la séquence de ce fragment ADN sont faibles entre les individus d’une même espèce et élevées entre les individus d’espèces différentes.
Le principal marqueur utilisé comme « code-barres » est un gène mitochondrial codant pour la principale sous—unité de la cytochrome oxydase, permettant l’identification d’une très grande partie du règne animal. Du fait que cette région évolue très lentement chez certaines espèces, elle ne permet pas toujours une identification, c’est pourquoi d’autres régions plus spécifiques ont également été retenues comme code-barres universels. Les gènes matK et rbcL par exemple, présent dans les chloroplastes, permettent une identification plus précise chez les plantes.
Le « code-barres » obtenu, associé au nom de l’espèce sur laquelle l’ADN a été prélevé, est ensuite inséré dans une base de données.
Gène rbcL : Code pour la grande sous unité de la Rubisco (ribulose-1,5-bisphosphate carboxylase /oxygénase), une protéine intervenant dans la fixation du carbone dans le cycle de Calvin. Durant ce cycle, l’énergie chimique acquise (ATP + NADPH) et stockée lors de la première étape de la photosynthèse, va être utilisée pour transformer le CO2 en composés organiques utilisables par l’organisme.
ITS (Internal Transcibed Spacer) : Région intergénique non codante encadrant l’ARN ribosomique 5.8S chez les eucaryotes. Elle est composée d’une courte séquence d’ADN répétée en tandem, c’est-à-dire à la suite l’une de l’autre, où le nombre de répétitions varie d’une espèce à l’autre. Du fait d’une plus grande variabilité que toutes autres régions, elle est désormais devenue une séquence universelle pour le barcoding.
Gène cytochrome oxydase I (COI) : Gène mitochondrial codant pour l’une des trois sous unités de la cytochrome c oxydase, une enzyme transmembranaire de type oxydoréductase. Elle est la dernière à intervenir dans la chaine respiratoire mitochondriale.
PROTOCOLE
Extraction d’ADN
Les spécimens utilisés pour l’extraction d’ADN sont : la drosophile (fournie par nos soins) et le champignon de Paris blanc ou brun (à acheter dans le commerce)
*Note: Pour les drosophiles : utiliser 1-2 mouches/tube, pour les champignons: un morceau d'environ 0.5cm3
-
Mettre 100 µl de NaOH 200mM dans un tube eppendorf.
-
Ajouter l'échantillon dans le tube et broyer à l’aide d’une pointe jaune. *
-
Fermer votre tube et incuber 10 minutes à 95°
-
Ajouter 100 µl de HCl 200mM
-
Ajouter 100 µl de Tris 200 mM
Votre préparation d’ADN est prête à être utilisée ou peut être stockée au congélateur.
Amplification par PCR
Préparer le mélange de réactifs pour la PCR juste avant l'emploi. Ce mélange contient le 2 x Taq mix (taq polymérases, dNTPs, tampon de polymérisation et tampon de charge rouge), les primers, le DMSO et de l’H2O.
Pour une classe de 16 élèves par exemple, on compte 3 réactions par binôme (un ADN, un contrôle négatif ainsi qu'un contrôle positif) soit un total de 24 réactions. Afin de faciliter le pipetage et pour avoir assez de matériel en cas d'erreurs nous prévoyons un mélange pour 28 réactions.
Nous allons ici effectuer une PCR multiplexe, c'est à dire que le mix PCR contient 4 primers. Un couple de primers spécifiques pour les champignons et un couple de primers spécifiques pour les insectes, permettant ainsi d'amplifier avec un seul mix l'un ou l'autre des ADN (ou les deux simultanément).
Champignons: amplification dans la région ITS
Insectes : amplification dans le gène codant pour la cytochrome oxydase
| 1 réaction | 28 réactions | |
| 2xTaqReadyMix | 12.5 µl | 350 µl |
| Primers mix | 8 µl | 224 µl |
| DMSO | 1.25 µl | 35 µl |
| H2O | 0.25 µl | 7 µl |
-
Ce mélange est préparé dans un tube Eppendorf 1.5 ml juste avant l’emploi. Mélanger en pipetant en haut et en bas délicatement.
-
Distribuer ensuite les 22 µl du mélange dans les petits tubes de PCR préalablement marqués sur le côté avec les initiales de l’élève.
-
Dans votre petit tube ajouter 3 µl de votre solution d’ADN. Fermer le tube.
-
Dans le petit tube PCR destiné au contrôle négatif, ajouter 3 µl d’H2O à la place de l’ADN. Fermer le tube.
-
Dans le petit tube PCR destiné au contrôle positif, ajouter 3 µl d’ADN c+. Fermer le tube.
-
En suivant les instructions du mode d'emploi du Bentolab, mettre les tubes dans la machine PCR et lancer le programme « BARCODING »
-
Le programme dure environ 1h40. Il comporte les cycles suivants :
| 94° 3 minutes | ||
| 94° 40 secondes | } | |
| 64° 40 secondes | 35 cycles | |
| 72° 1 minute 30 | ||
| 72° 3 minutes | ||
| 4° pause |
Préparation du gel d'agarose
Le TAE est toxique pour l’environnement, il ne faut donc pas le jeter à l’évier. Celui-ci doit être récupéré et éliminé par le biais de votre propre filière de traitement des produits chimiques.
Le gel d'agarose est une matrice pour séparer les molécules d'ADN selon leur taille dans un champ électrique. Les petits fragments migrent plus vite que les grands, la concentration d'agarose est choisie en fonction de la taille des fragments à séparer. Ici nous allons utiliser un gel à 1.5 % d’agarose.
-
Peser 0,75 g d'agarose et mettez-le dans un Erlenmeyer de 250ml.
-
Ajouter 50 ml de tampon d'électrophorèse (TAE 1x).
-
Faire bouillir (four à micro-ondes, plaque chauffante ou bec bunsen). Veillez à ce que l’agarose ne déborde pas en chauffant ! L’agarose fondu est complètement transparent. Au cas où des résidus sont toujours visibles, remettre la solution à chauffer.
-
Laisser refroidir le liquide (à environ 60°C).
-
Ajouter 5 µl de SYBR-safe (intercalant qui permettra de visualiser l’ADN) et mélanger en agitant l’Erlenmeyer.
-
Verser l'agarose dans la cuve préparée avec les protections pour les électrodes et un peigne pour former les puits (cf. image)
- Lorsque le gel est refroidi et solidifié, retirer les protections, ajouter du tampon d'électrophorèse (TAE 1x) et enlever le peigne.

Analyse des résultats sur gel
-
Récupérer vos tubes. Ils contiennent déjà le tampon de charge (rouge) et sont donc prêts à être chargés dans les puits.
-
Charger le gel :
1er puits : 5 µl de marqueur.
2ème puits et suivants : 20 µl de vos échantillons.
Avant dernier puits : le contrôle négatif.
Dernier puits : le contrôle positif.
-
Mettre le couvercle, brancher les électrodes et lancer la migration du gel.
-
Quand le colorant rouge de migration arrive en bas du gel, arrêter le transformateur.
-
Laisser refroidir le gel quelques instants. Eventuellement en le mettant au frigo. L’intensité du signal sera ainsi plus forte.
-
En lassant le gel dans la cuve, allumer la lampe bleue pour visualiser les résultats.
-
Prendre une photo si nécessaire.
Note: Si l'intensité des bandes n'est pas suffisante, poser le gel directement sur la lampe bleue.
Exemple de résultats:
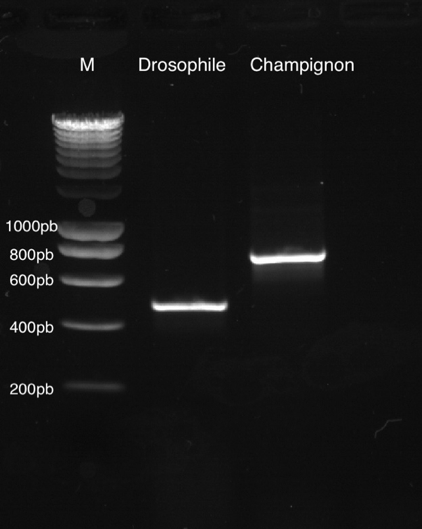
Matériel fourni:
- Bentolab
- 2x Taq ReadyMix
- Primers mix
- DMSO
- ADN contrôle
- Marqueur de taille (électrophorèse)
- Solution de SYBR-safe
- NaOH 200mM
- HCl 200mM
- Tris-HCl 200mM
- Micropipettes P20
- Micropipettes P200
- Micropipettes P1000
- Boîte de pointes jaunes
- Boîte de pointes bleues
- Bloc chauffant
- Boite tubes PCR
- Boite tubes Eppendorf 1,5 ml
- Tampon d'électrophorèse (TAE 1x)
- Agarose
- Bouteille d'eau stérile
Matériel non fourni:
L’Université de Genève décline toutes responsabilités en cas de dommages survenus durant les expériences.

