Le clonage moléculaire est une des bases du génie génétique. Il consiste à insérer un fragment d'ADN (dénommé insert) dans un vecteur approprié comme un plasmide par exemple (voir exp4 pour une explication sur les plasmides). Le nouveau plasmide ainsi créé sera ensuite introduit dans une cellule hôte, en générale la bactérie Escherichia coli. Celle-ci sera alors sélectionnée et multipliée afin d’obtenir une grande quantité du plasmide d’intérêt. Cloner un gène consiste donc à l’insérer dans un plasmide. Un clone sera le transformant bactérien qui contient ce plasmide particulier. Dans ce cas on parle de clone car tous les individus de la colonie bactérienne sont génétiquement identiques. Le clonage moléculaire est donc différent du clonage reproductif (créer un individu génétiquement identique à un autre mais d'un age différent) ou du clonage thérapeutique (fabriquer des tissus à partir de cellules souches pour effectuer des greffes compatibles avec le receveur).
Thèmes: clonage moléculaire, vecteurs, hôtes, digestion, ligation, transformation bactérienne, X-gal
Le clonage moléculaire nécessite des enzymes de restrictions capables de couper l’ADN (exp2), et de l’ADN ligase capable de recoller les fragments d’ADN. La ligase a été isolée pour la première fois à partir du bactériophage T4. Cette enzyme est impliquée dans la réparation et la réplication de l’ADN. Elle peut lier des fragments d’ADN ayants des bouts collants (exp2) compatibles. A plus grande concentration, cette enzyme est également capable de lier deux bouts francs d’ADN comme indiqué ici. La T4 ADN ligase fonctionne en utilisant de l’ATP et du Mg++. Elle possède un optimum d’activité de 16°C, mais reste bien active à température ambiante.
Le premier clonage fut réalisé en 1972 par Paul Berg qui partagea le prix Nobel de chimie en 1980 avec Walter Gilbert et Frederick Sanger. En 1977, le premier gène humain, codant pour la somatostatine est cloné permettant aux bactéries de produire des protéines humaines. L’ère du génie génétique et des biotechnologies venait alors de démarrer.
L’EXPERIENCE : (Schéma de l’expérience)
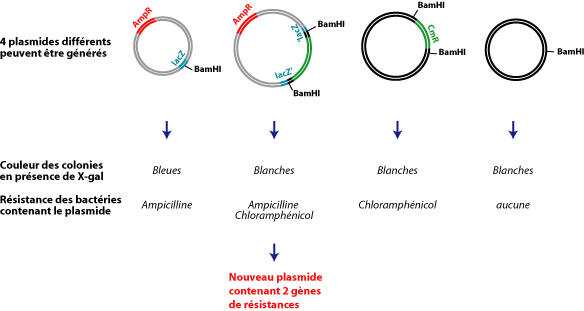
Dans cette expérience nous allons utiliser deux plasmides portant des marqueurs de résistances aux antibiotiques différents. Le plasmide récepteur (pUC19) contient le gène de résistance pour l’antibiotique ampicilline. Il sera linéarisé par l'enzyme de restriction BamHI. Le plasmide "donneur" pHP45 porte un fragment contenant le gène codant pour la résistance au chloramphénicol. Il sera coupé en 2 parties par l’enzyme de restriction BamHI libérant ainsi le gène de résistance au chloramphénicol. Le vecteur pUC19 linéarisé et le produit de digestion de pHP45 seront ensuite mélangés. La ligase va permettre la formation d’un nouveau plasmide contenant les deux gènes de résistances pour l’ampicilline et le chloramphénicol. Après transformation du produit de la ligation dans E. coli, seules les bactéries qui ont incorporé ce plasmide pourront pousser sur un milieu de culture contenant les 2 antibiotiques. Ces bactéries seront donc facilement sélectionnées.
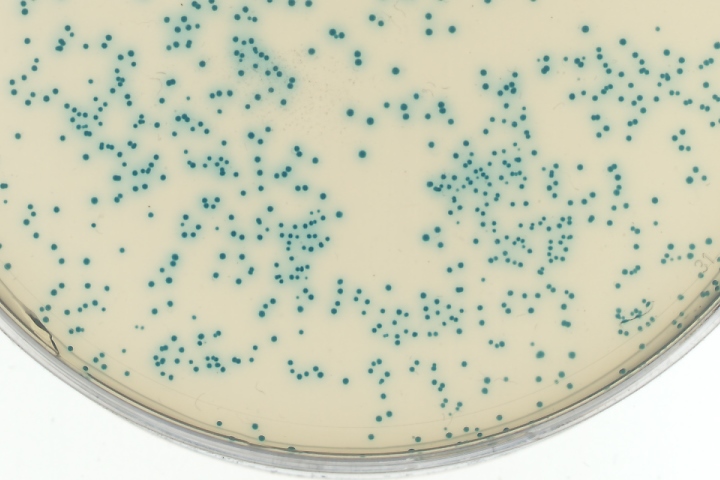
Les fragments à cloner ne sont pas toujours aussi simple à repérer. C'est pourquoi beaucoup de vecteurs portent un petit bout du gène lacZ (codant pour la β-galactosidase). Cette enzyme transforme le lactose, un disaccharide, en deux monosaccharides, le galactose et le glucose. Cette enzyme transforme également le X-galactose (X-gal) incolore en un produit bleu insoluble qui s’accumule dans les cellules. Si un fragment d'ADN est inséré dans le gène lacZ, celui-ci ne sera plus fonctionnel et la conversion du X-gal ne sera plus possible. Les colonies resteront blanches. En absence d’insert, le gène lacZ sera fonctionnel et la colonie bactérienne deviendra bleue. Cette sélection bleu/blanc est un moyen très pratique de sélectionner les colonies qui contiennent le plasmide avec insert.
Protocole:
1) Digestion
Les plasmides pUC19 (carte de restriction, voir exp2) et pHP45-CmR vont être coupés avec l’enzyme de restriction BamHI (volumes en µl, total 20 µl):
- Numéroter 2 tubes Eppendorf
- Ajouter les éléments comme décrit dans le tableau ci-dessous (les volumes sont en µl).
| H2O | pUC19 | pHP45Cm |
Tampon de digestion 10x |
BamHI | |
| Tube 1 | 13 µl | 4 µl | - | 2 µl | 1 µl |
| Tube 2 | 9 µl | 8 µl | 2 µl | 1 µl |
Attention : Garder l'enzyme de restriction sur glace - Utiliser une pointe neuve pour chaque pipetage
- Mélanger en pipetant plusieurs fois.
- Incuber la réaction à 37°C de 30 à 60 minutes.
- Placer les tubes à 70°C 15 minutes pour inactiver l'enzyme de restriction.
2) Ligation
- Prendre 2 nouveaux tubes marqués L (pour ligation) et C (pour control).
- Ajouter les éléments comme décrit dans le tableau ci-dessous (les volumes sont en µl).
| Tube 1 | Tube 2 | H2O | Tampon de ligation | DNA ligase | |
| Tube L | 3 µl | 10 µl | 4 µl | 2 µl | 1 µl |
| Tube C | 3 µl | 10 µl | 5 µl | 2 µl | - |
Attention : Garder la ligase sur glace - Utiliser une pointe neuve pour chaque pipetage
- Laisser les tubes de 15 à 30 minutes à température ambiante pour permettre la ligation des fragments d'ADN.
3) Transformation
Les bactéries Escherichia coli sont rendues compétentes comme décrit dans l'expérience 4.
- Ajouter 10 µl du tube L dans 100 µl de cellules compétentes. Marquer ce tube TL (transfo L).
- Ajouter 10 µl du tube C dans 100 µl de cellules compétentes. Marquer ce tube TC.
- Ajouter 10 µl d'H2O dans 100 µl de cellules compétentes (controle négatif de transformation. Marquer ce tube T-.
- Mettre les tubes 15 minutes sur glace
- Incuber les tubes 2 minutes à 42°C (choc thermique)
- replacer les tubes 5 minutes sur glace
- Ajouter 1 ml de LB dans chaque tube et incuber la suspension à 37°C pendant 15 à 30 minutes (pour l’expression de la résistance à l’ampicilline).
- Prélever de chaque tube:
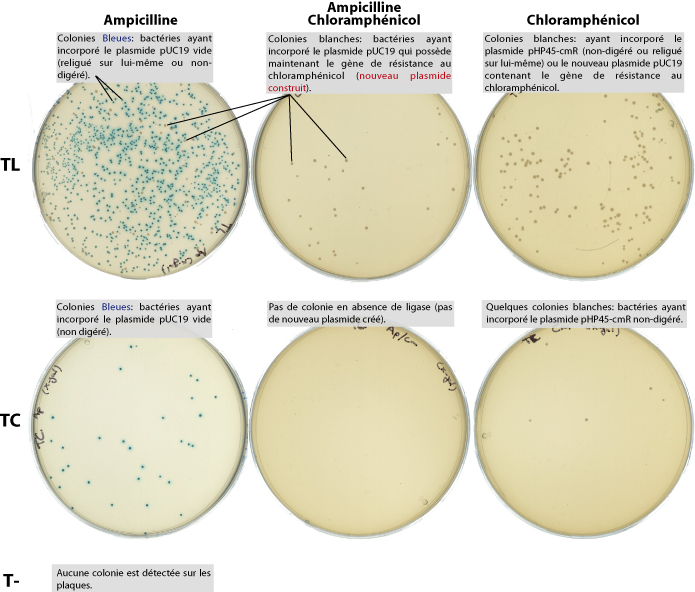
- 0.1 ml et étaler sur une boîte LA +ampicilline +X-gal
- 0.1 ml et étaler sur une boîte LA + ampicilline+ chloramphénicol + X-gal
- 0.1 ml et étaler sur une boîte LA + chloramphénicol + X-gal
- Marquer les boîtes sur le fond avec votre nom et le numéro du tube de transformation
- Incuber les boîtes à l'envers à 37°C pendant une nuit. les boîtes peuvent ensuite être gardées au frigo (4°C).
4) Analyse des résultats
Le schéma ci-dessous nous montre les 4 plasmides que l'on peut obtenir et les résultats attendus sur boîtes.
Sur boîtes de pétri voila ce que l'on obtient:
Matériel fourni:
- Micropipettes P20
- Micropipettes P200
- Boîte de pointes jaunes
- Bloc chauffant
- Boite tubes Eppendorf 1,5 ml
- Portoir tube eppendorf
- Bouteille d'eau stérile
- Boîtes de Petri LA + X-Gal + chloramphenicol
- Boîtes de Petri LA + X-Gal + ampicilline + chloramphenicol
- Plasmide pUC19
- Plasmide pHP45-cmR
- Enzyme de restriction BamHI
- Tampon de digestion
- ADN ligase
- Tampon de ligation
- Cellules compétentes Escherichia coli
- Bloc réfrigérant
- Boîte pipettes Pasteur pour les étalements
- Boîtes de Petri LA + X-Gal + ampicilline
- Milieu LB liquide
- Sac jaune pour les déchets
Matériel non fourni:
L’Université de Genève décline toutes responsabilités en cas de dommages survenus durant les expériences.